仕事で使ったら、どっぷり浸かってしまったRです。
もちろん、得手不得手はあるのですが、アイデアが浮かんだらちょいちょいと書いてみて結果を確認するとか、取得したデータをざっと見て今後の解析の方針を考えるとか言うのに最適です。
もちろん、スクリプト言語ですから、定型作業の簡素化にも、役立ちます。
基本的に数字をいじるのに使うソフトですが、画像だってデジタルデータですから、画像処理に使ったりする人までいます。
Rについては、一昨年、授業の実習でよく使っていました。
講師の先生曰く、自分で書かなくても、捜せばどこかに誰かが書いたプログラムがあるので、読み込む、または、copy&paste(コピーして貼付け)して使いなさい。
Rでプログラムをすること自体が目的でなくて、自分の手元のデータを解析して結果をだすことが目的なのだから。
わたしは、その教えを忠実に守ることにしています(w
ここ数日の周期解析の話も、Katさんが書かれたプログラムをいじっているだけです。
勿論、解析にあたっては原理等を理解することは大切で、自分が、今、何をやっているかを理解することは基本です。
でも、copy&patsteしてるだけでも、その辺りは、心がけていれば、何となく身に付いていくものです。
さて、ここ2-3日、Katさんの書かれた、Rによる周期解析ソフトをいじってみて、これは、使えるという印象を持ちました。
Rになじみの無い方にも使って欲しいので、Windowsを例に、まとめてみます。
Rが初めての方のために、最初に、ごちゃごちゃ書きました。周期解析が早くしたという方は、読飛ばしてください。
なお、以下の解説は、Windows7を例にしています。
#OSXや、Linuxを使っている方は、適宜、読み変えて、わからないとこはgoogleって調べましょう。
(Rについて)
Rそのものについては、wikipediaを参考にしてください。
installの仕方は、googleと沢山出てきます。
例えば、このあたり(R のセットアップ+ R 入門 続・わしの頁)
図もあるので、慣れていない方でも、この通りやれば、デスクトップにRのアイコンが出来ます。
バージョンは、今は、 2.13.1です。
これをダブルクリックするとR用のGUIが立ち上がると思います。
ある程度の設定が、GUIで出来ますが、基本は、>のプロンプトの後にコマンドを打っていきます。
インタプリタ型なので,結果がすぐ表示されて、半対話的に解析が行えます。
また、そのためcopy&pasteとの相性が良いです。
# 解析がうまくいったら、その手順をcopy&pasteしてファイルに保存しておけば、後から、再利用が出来ます。
区切りのためのスペースは、基本的には無視されるので、読み易い様に打ち込めます。また、単語の区切りに合わせれば行の途中のどこでも改行が出来きます。
そのため、1行が長くなる時は、リターンを押すと
+
が表示されて、続きが入力できます。
使い方が、わからない時は、help(plot)の様に、知りたいコマンド名をいれれば、英語ですが、マニュアルが表示されます。
Rで一番特徴的なのは、代入に <- を使うことです。=でも代入できるのですが、<-を多用した方が、Rを使っている気分が出ます。
=は、主に、パラメータを指定する時に使います。
スカラー型、ベクター型のデータを扱うことが出来、行列として扱うことも出来ます。
あと、特徴的なのは、文字と数字が混在したデータを扱えることで、data.frameと呼ばれています。
ここらあたりもwikipediaの解説に詳しいです。
(DFTによる変光星の周期解析)
ここからは、how to的に、操作を書いていきます。逆に、原理とかの説明は、別途とさせてください。
まず、準備です。
観測データは、Katさんのつくられた、dbvs2000のシステムを利用します。
ファイルは、ここにあるので、適当なフォルダーの中に解凍してください。
データファイルは、ここです。解凍すると、vsolj.dbfとvsolj.ctlという2つのファイルが入っています。
これも、dbvs2000と同じフォルダーに入れておきます。
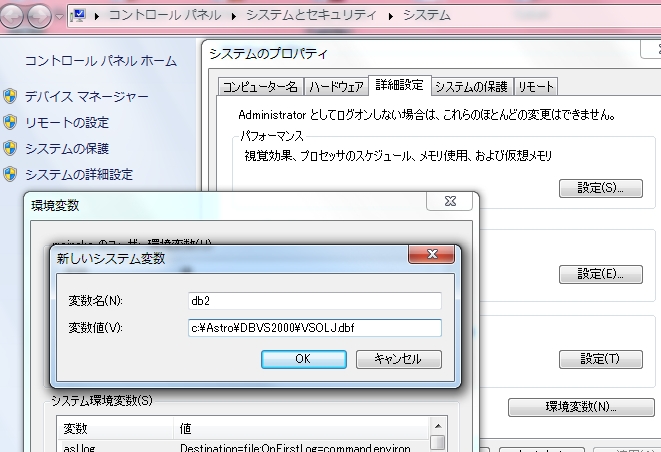
このvsolj.dbfの在処をdbv2000システムに教えるために、環境変数を設定しておきます。
Windowsメニューのコンピュータを右クリックして、プロパティを選ぶと、コントロールパネルのシステムが表示されます。
左に、システムの詳細設定を選で表示されたシステムのプロパティの環境設定を押します。
システム環境設定で、新規を選んで、変数名にdb2、変数値に先ほどのdbvs2000のフォルダーを指定します。
ちなみに、わたしの場合は、c:\astro\dbvs2000\vsolj.dbfです。
Rの他に、awkというスクリプト言語も使うので、これをあらかじめinstallして、どこからでも実行できる様にpathを通してしておきます。
#このあたり、最初から入っているOSXやLunixと違って不便です。
例えば、ここから。
なお、awkは、複数の方が、Windows用に移植しているので、名前が、gawkとかになっているかもしれません。
GNU awkのことで、FSFのGNU projectで移植されたフリーウェアです。以下、gawkを前提にして書きます。
コマンドプロンプトを立ち上げて、dbvs2000のフォルダーに移動して、gwakと打ち込んでみて、使い方の説明がザーと表示されたらOKです。
pathの指定が良くわかなければ、gawk.exeもdbvs2000のフォルダーにcopyしてしまうのも、緊急措置的にはありかもしれません。
#なお、gawkだけ単独で入れるのではなく、gnuwin32とかcygwinとかを使って、unix関連のソフトを一度に入れてしまうのも手です。
#方法は、googleて下さい。インストーラーが用意されているので、ファイルを落としてダブルクリックでするだけで、あとは、インストーラーの指示通りでインストールできるとは思います。
#このあたり、最初から入っているOSXやLunixと違って不便です。しつこい。
ちなみに、コマンドプロンプトで、カレントフォルダーの変更は例えば、
cd c:\astro\dbvs2000
こんな感じです?
コマンドプロンプトは、メニュー>アクセサリの中です。
次に、awkから呼び出して使うjdfil.awkというファイルを作ります。
awkに仕事の内容を指定するファイルなのですが、長いので、ファイルにして保存しておいて使います。
以下をメモ帳かなにかにcopy&pasteして、jdfil.awkという名前で、dbvs2000のフォルダーに保存します。
BEGIN { } { if (substr($0,1,1) != "#" && index($2,"<") == 0) { mag = $2; gsub("[<A-Za-z:]","",mag) if (index(mag,".") == 0) { mag = sprintf("%.1f",mag*0.1) } printf("%s %s\n",$1,mag) } }
以上で、下ごしらえは終わりです。
さて、Rを上げて、ファイル>ディレクトリの変更から、dbvs2000のフォルダーを指定します。
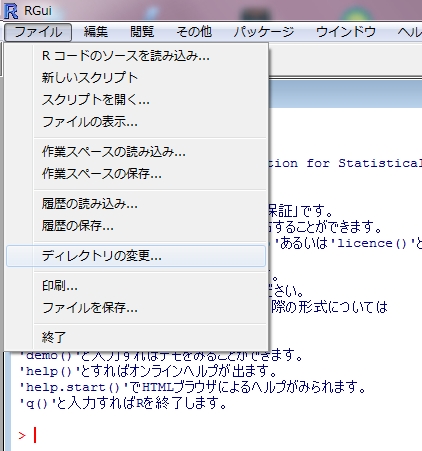
プログラムは、いくつかに分かれていて、
readdb
lfit
pergrm
max
phave
等から、なっていますが、以下をcopyして、Rにpsteしてやるだけです。
readdb <- function(obj,start="19000101",end="20991231") { cmd <- sprintf("vcut %s %s-%s | pack | tojd | gawk -f ./jdfil.awk > temp.txt",obj,start,end) system(cmd) read.table("temp.txt") } pergrm <- function(d,start,end,div) { d$V2 <- d$V2 - mean(d$V2) p <- seq(start,end,length=div+1) pw <- as.numeric(lapply(p, function(tp) { ph <- ((d$V1/tp) %% 1)*pi*2 (sum(d$V2*sin(ph)))^2 + (sum(d$V2*cos(ph)))^2})) r <- data.frame(period=p,power=pw) class(r) <- "Pergrm" return(r) } plot.Pergrm <- function(pgm,ylim=NULL,xlab="Period",ylab="Power",...) { if (is.null(ylim)) { ymax <- max(pgm$power) ymin <- min(pgm$power) y1 <- ymin - (ymax-ymin)*0.05 y2 <- max(1,ymax + (ymax-ymin)*0.05) yl <- c(y1,y2) } plot(x=pgm$period, y=pgm$power, xaxs="i", ylim = yl, typ="l", xlab = xlab, ylab = ylab, ...) } max.Pergrm <- function(pgm,ma.rm=FALSE,...) { pgm$period[which.max(pgm$power)] } lfit <- function(dat) { res <- lm(dat$V2 ~ dat$V1) dat$V3 <- dat$V2 dat$V2 <- res$residuals return(dat) } phase <- function(dat,epoch,period) { if (class(period) == "minper") { p <- period$minper[1] } else { p <- period } f <- dat f$mag <- dat$V2 f$ph <- ((dat$V1 - epoch) %% p)/p if (!is.null(dat$err)) { f$err <- dat$err } f1 <- subset(f,f$ph < 0.5) f1$ph <- f1$ph + 1 f2 <- subset(f,f$ph >= 0.5) f2$ph <- f2$ph - 1 f <- rbind(f2,f,f1) class(f) <- "phase" return(f) } phave <- function(dat,epoch,period,ndiv=60) { if (class(period) == "minper") { p <- period$minper[1] } else { p <- period } dat$ph <- ((dat$V1 - epoch) %% p)/p br <- seq(0,1,length=ndiv+1) dat$bin <- factor(cut(dat$ph,breaks=br,labels=FALSE)) ph <- as.vector(tapply(dat$ph,dat$bin,mean)) num <- as.vector(tapply(dat$V2,dat$bin,length)) mag <- as.vector(tapply(dat$V2,dat$bin,mean)) err <- as.vector(tapply(dat$V2,dat$bin,sd)) err <- ifelse(num > 1, err/sqrt(num-1), NA) f <- data.frame(ph,mag,err,num) f1 <- subset(f,f$ph < 0.5) f1$ph <- f1$ph + 1 f2 <- subset(f,f$ph >= 0.5) f2$ph <- f2$ph - 1 f <- rbind(f2,f,f1) row.names(f) <- 1:nrow(f) class(f) <- c("phave",class(f)) return(f) } plot.phave <- function(a,xlim=NULL,ylim=NULL,xlab="Phase",ylab="Mag",axes=TRUE) { if (is.null(ylim)) { ymax <- max(a$mag) ymin <- min(a$mag) y1 <- ymin - (ymax-ymin)*0.1 y2 <- ymax + (ymax-ymin)*0.1 yl <- c(y2,y1) } else { yl <- ylim } if (is.null(xlim)) { xlim <- c(-0.5,1.5) } plot(x=a$ph, y=a$mag, xlim = xlim, ylim = yl, pch = 19, xlab = xlab, ylab = ylab,axes=axes) arrows(a$ph, a$mag-a$err, a$ph, a$mag+a$err, length=.05,angle=90,code=3) } phave.save <- function(a,outfile) { fout <- file(outfile,"w") for (i in 1:nrow(a)) { s <- sprintf("%8.5f %8.5f %8.5f %4d\n", a$ph[i],a$mag[i],a$err[i],a$num[i]) cat(file=fout,s) } close(fout) } yerr.plot <- function(x,y,e,ylim=NULL,xlab="x",ylab="y") { plot(x=x, y=y, pch = 19, ylim=ylim, xlab = xlab, ylab = ylab) arrows(x, y-e, x, y+e, length=.05,angle=90,code=3) } plot.phase <- function(a,xlim=NULL,ylim=NULL,xlab="Phase",ylab="Mag",axes=TRUE) { if (is.null(ylim)) { ymax <- max(a$mag) ymin <- min(a$mag) y1 <- ymin - (ymax-ymin)*0.1 y2 <- ymax + (ymax-ymin)*0.1 yl <- c(y2,y1) } else { yl <- ylim } if (is.null(xlim)) { xlim <- c(-0.5,1.5) } plot(x=a$ph, y=a$mag, xlim = xlim, ylim = yl, pch = 19, xlab = xlab, ylab = ylab,axes=axes) } phave.points <- function(a,xlab="Phase",ylab="Mag") { points(x=a$ph, y=a$mag, pch = 19, xlab = xlab, ylab = ylab) arrows(a$ph, a$mag-a$err, a$ph, a$mag+a$err, length=.05,angle=90,code=3) }
さて、R側の準備も終わりました。
Rで、>の後に以下のように入力して、解析したい星の名前を、starに代入します。
#赤字が入力
star <- "CYGchi"
#入力が終わったら、リターンです。
vsolj.dbfからデータを読み込みます。
d <- readdb(star)
DFTで、周期解析をします。
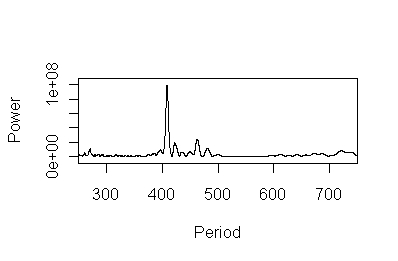
pgm <- pergrm(d,250,750,5000)
上の例では、250日が解析開始の周期、750日が終了の周期、その間を5000に分けて周期解析をしてます。
#この例だと、(750-250)/5000だから、0.01日刻みですね。
グラフを描いてみましょう。
plot(pgm)
ピークのところの周期をpに代入して、数字を確認します。
p <- max(pgm)
p
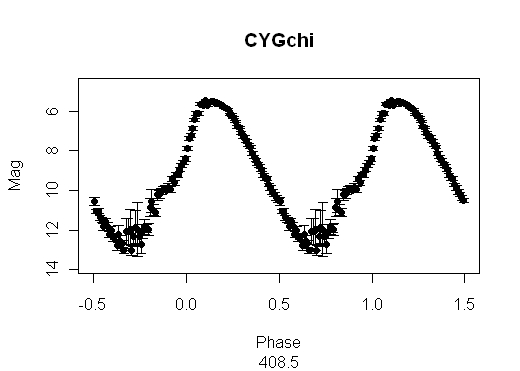
[1] 408.5
この周期を使って、位相を計算して、平均光度曲線を描いてみましょう。
plot(phave(d,0,p,100))
title(main=star, sub=p)
はい、出来ました。
なお、データを読み込む時に、
d <- readdb(star, 19900101, 20041231)
の様に、期間を指定することもできます。
#自宅のメインのPCで動かそうとしたら、Windows7が64ビットなので、dbvs2000が動かなかったですorz
#急遽、XPモードにRとgawkを入れるハメにorz